网站建设都需要买什么东西品牌全网推广
这里写目录标题
- 1.给出一个线性回归模型并求出因子贡献度
- 2.biomod2
- 2.1 pseudo-absences:伪不存在点(PA)
- 2.1.1 random
- 2.2.2 disk
- 2.2.3 user.defined method
- 3.使用网格划分区域
- 3.1 计算质心
- 4. 完整案例
1.给出一个线性回归模型并求出因子贡献度
##-----------------------------------------------------------------------------
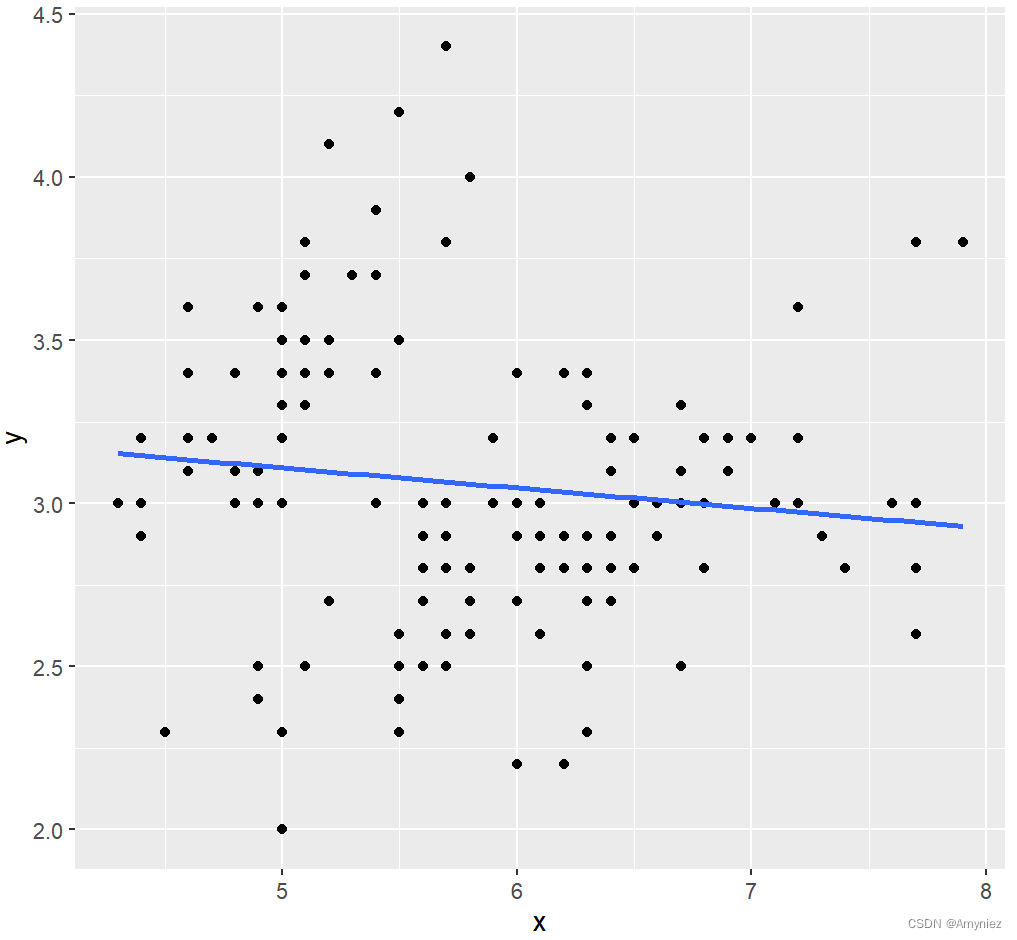
# 线性回归模型
lm_df <- data.frame(x = iris$Sepal.Length,y = iris$Sepal.Width)
lm_model <- lm(data = lm_df,y ~ x)
broom::tidy(lm_model)ggplot(data = lm_df,aes(x,y))+geom_point()+geom_smooth(method = lm, se = FALSE)# 变量重要性
install.packages("vip")
install.packages('mlbench')
library(vip)
set.seed(100)
trn <- as.data.frame(mlbench::mlbench.friedman1(500))
linmod <- lm(y ~ .^2, data = trn)
backward <- step(linmod, direction = "backward", trace = 0)
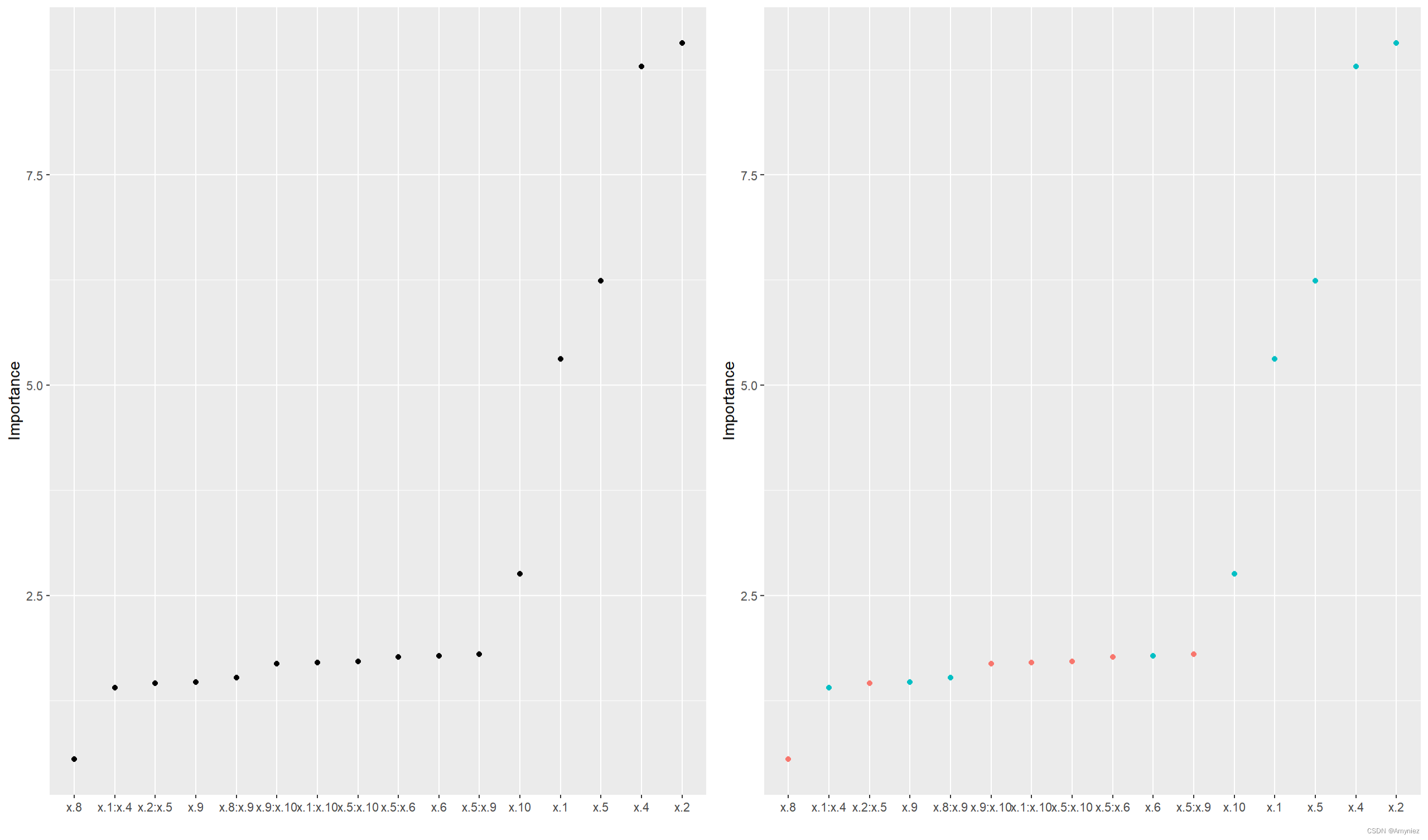
# 计算贡献度
vi(backward)# 可视化
p1 <- vip(backward, num_features = length(coef(backward)), geom = "point", horizontal = FALSE)
p2 <- vip(backward, num_features = length(coef(backward)), geom = "point", horizontal = FALSE, mapping = aes_string(color = "Sign"))
grid.arrange(p1, p2, nrow = 1)
结果展示:

图像绘制:
重要性结果展示:
2.biomod2
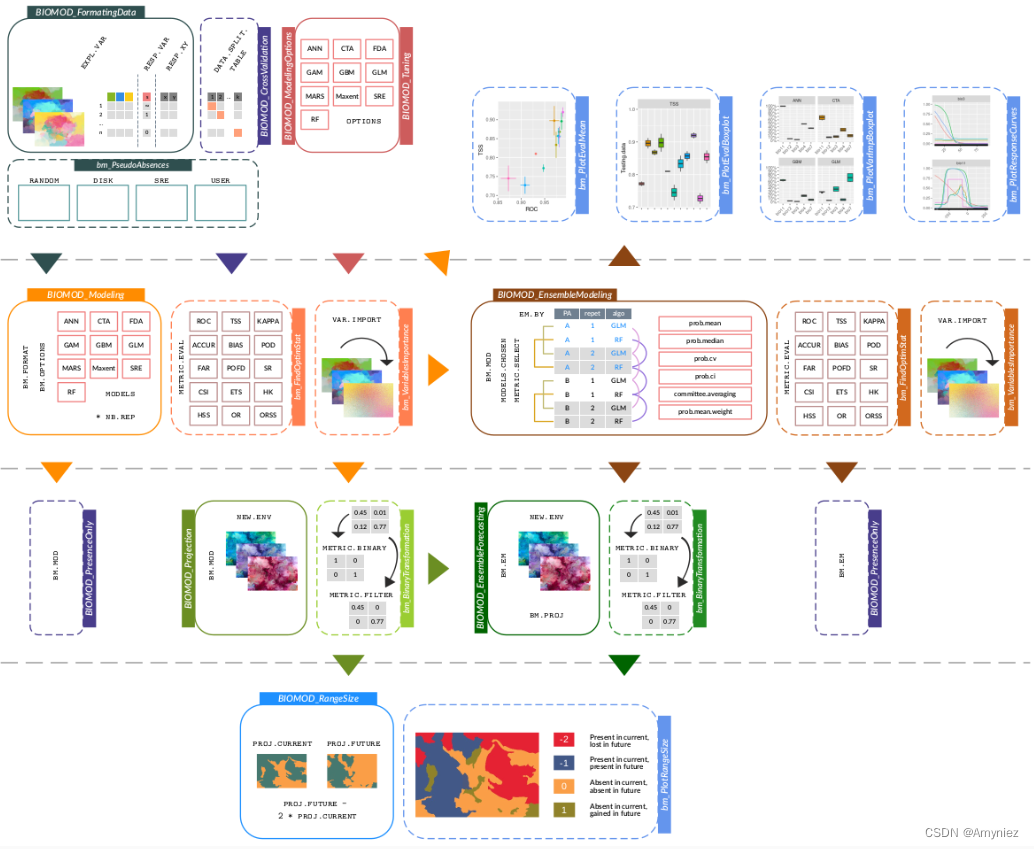
首先需要安装biomod2包:install.packages(“biomod2”)
最终生成的文件为individual_projections,该文件夹中包括.img、.xml两种数据格式,其中包括很多算法如,GLM,RF,SRE,ANN,CTA,FDA,CTA等多种模型 ,这类似于一个集成算法,集合多个模型,求取模型的平均值,以得出一个更好的模型。
##-----------------------------------------------------------------------------
# 加载成都市的适量边界图,后面会用到
library(mapchina)
cd_sf <- mapchina::china %>%dplyr::filter(Name_Perfecture == "成都市") %>%group_by(Name_Province) %>%summarise(geometry = sf::st_union(geometry)) %>%ungroup()
colnames(cd_sf) # see all variable names
plot(cd_sf)#install.packages("biomod2")
library(biomod2) ?biomod2::BIOMOD_FormatingData()
#
2.1 pseudo-absences:伪不存在点(PA)
生成PA点的四种方法:random、disk、sre、user.table
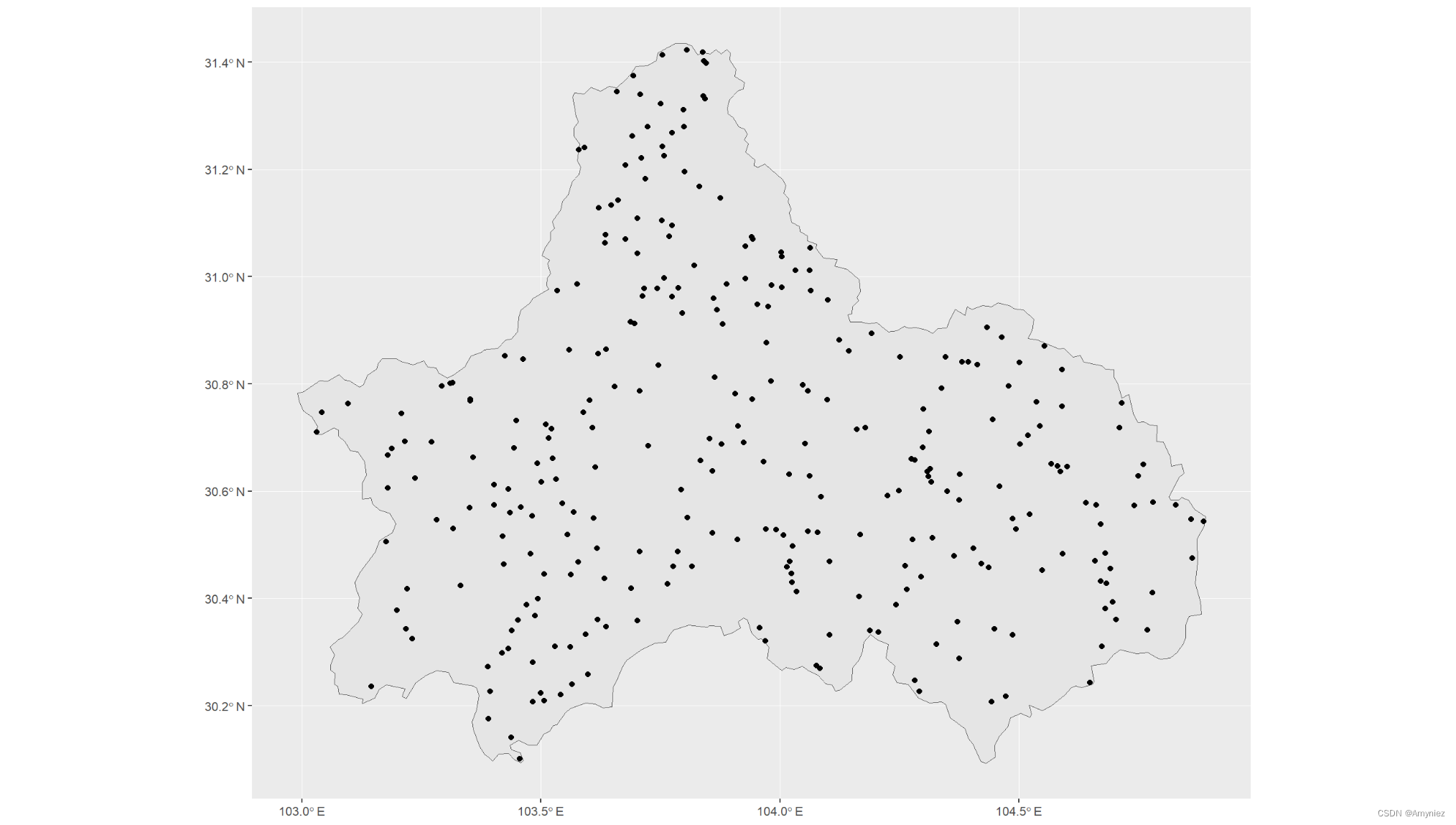
2.1.1 random
随机选择PA点
##--------------------------------------------------------------------------------
# 1.the random method : PA are randomly selected over the studied area (excluding presence points)
library(sf)
p1_random <- sf::st_sample(cd_sf,300)
ggplot()+geom_sf(data = cd_sf)+geom_sf(data = p1_random)
结果展示:
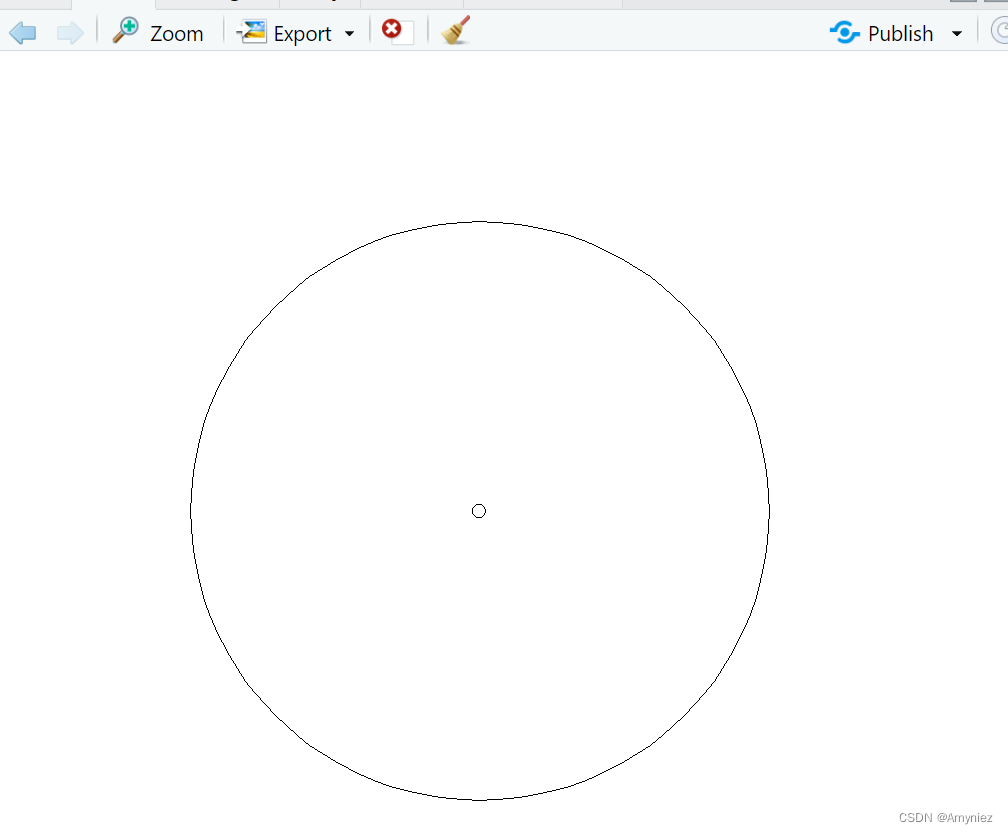
2.2.2 disk
# 2.the disk method : PA are randomly selected within circles around presence
# points defined by a minimum and a maximum distance values (defined in meters).
## Format Data with pseudo-absences : disk method
# myBiomodData.d <- BIOMOD_FormatingData(resp.var = myResp.PA,
# expl.var = myExpl,
# resp.xy = myRespXY,
# resp.name = myRespName,
# PA.nb.rep = 4,
# PA.nb.absences = 500,
# PA.strategy = 'disk',
# PA.dist.min = 5,
# PA.dist.max = 35) # 生成环形缓冲区
pts_presence <- sf::st_sample(cd_sf,300)
pts_presence

#使用生成的第一个点画圆
st_buffer(pts_presence[[1]], dist = 1) %>% plot()
plot(pts_presence[[1]],add = TRUE)
结果展示:
2.2.3 user.defined method
##-------------------------------------------------------------------------------------
#用户自定义
## Format Data with pseudo-absences : user.defined method
# myPAtable <- data.frame(PA1 = ifelse(myResp == 1, TRUE, FALSE),
# PA2 = ifelse(myResp == 1, TRUE, FALSE))
# for (i in 1:ncol(myPAtable)) myPAtable[sample(which(myPAtable[, i] == FALSE), 500), i] = TRUE
# myBiomodData.u <- BIOMOD_FormatingData(resp.var = myResp.PA,
# expl.var = myExpl,
# resp.xy = myRespXY,
# resp.name = myRespName,
# PA.strategy = 'user.defined',
# PA.user.table = myPAtable)
pts_absence <- pts_presence %>% st_as_sf() %>% mutate(id = 1:n()) %>%group_by(id) %>%nest(data = -id) %>% mutate(circle = purrr::map(.x = data,.f = function(x) {st_buffer(x = x,dist = 1)})) %>% mutate(point = purrr::map(.x = circle,.f = function(x) {st_sample(x,1)})) %>% dplyr::select(point) %>% unnest() %>% ungroup() %>% dplyr::select(-id)
pts_absence

格式转换:
# 将生成的点转换为数据框格式
#install.packages('sfheaders')
library(sfheaders)
pts_absence %>% st_as_sf() %>% sfheaders::sf_to_df() %>% dplyr::select(x,y) %>% mutate(label = "absence") %>% head()

3.使用网格划分区域
##----------------------------------------------------------------------------------
# 网格划分,形成栅格图像
cd_grid <- cd_sf %>% st_make_grid(cellsize = 0.2) %>%st_intersection(cd_sf) %>%st_cast("MULTIPOLYGON") %>%st_sf() %>%mutate(cellid = row_number())
plot(cd_grid)
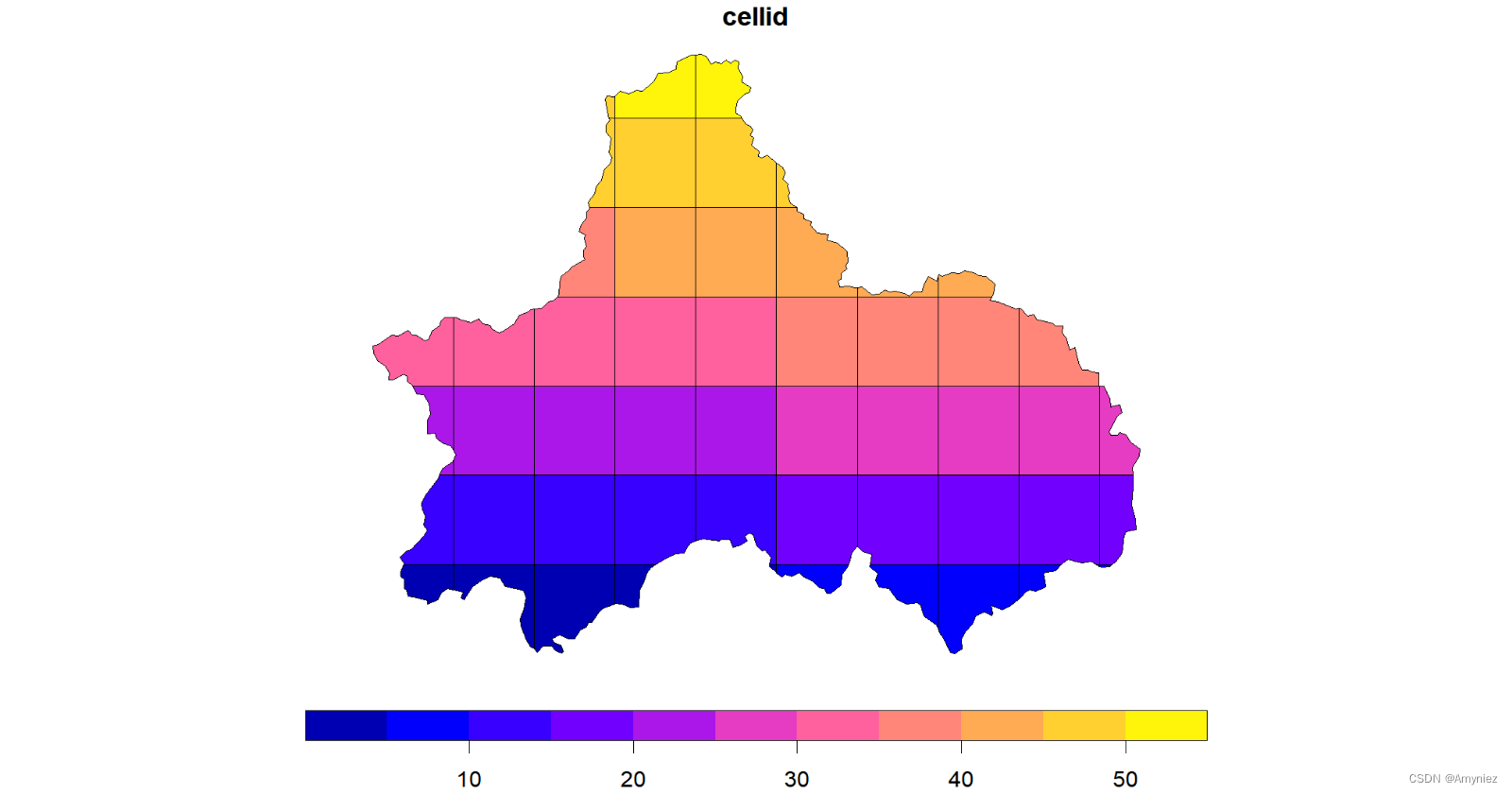
为每个网格添加标签:
#devtools::install_github("yutannihilation/ggsflabel")
ggplot(data = cd_grid)+geom_sf()+ggsflabel::geom_sf_label(aes(label = cellid))+theme_light()
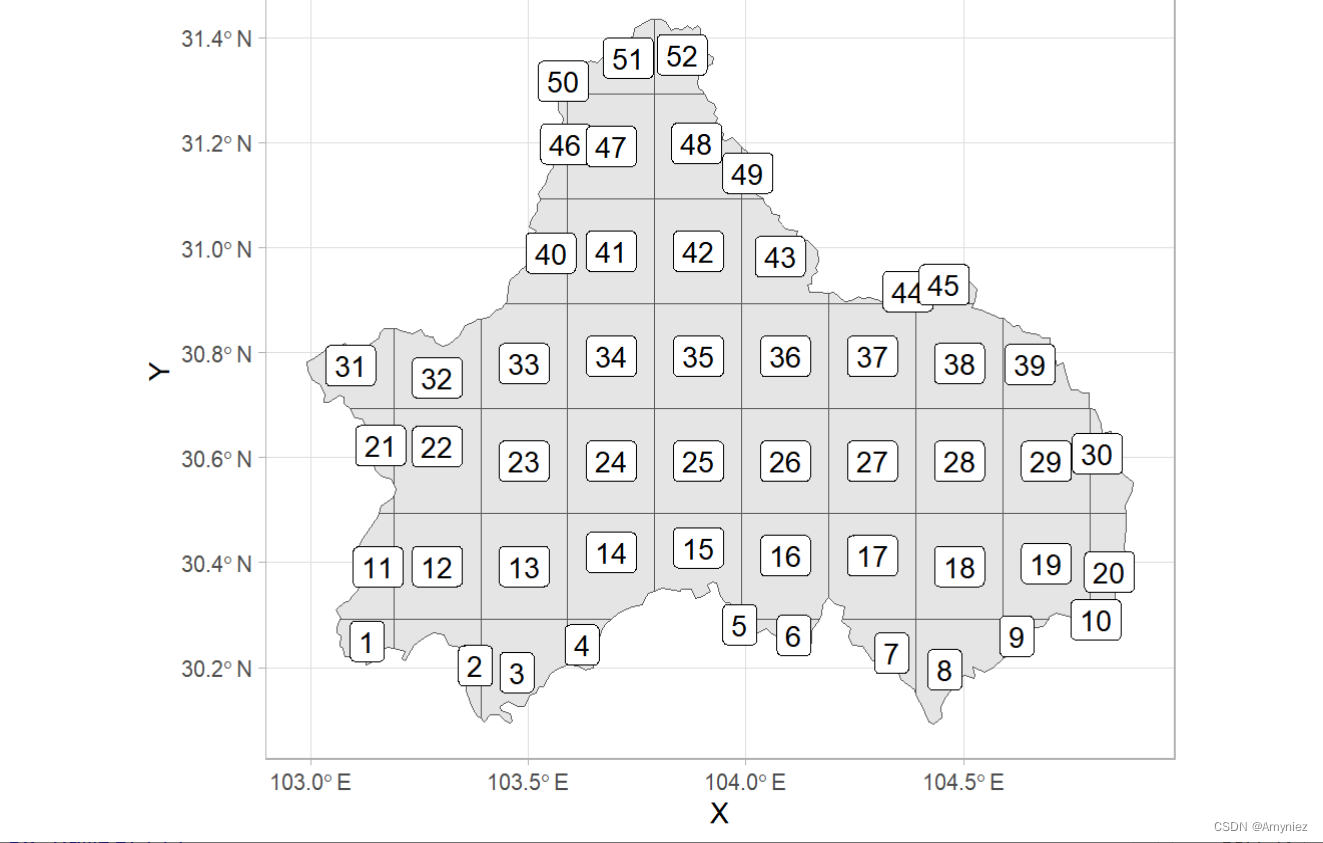
3.1 计算质心
# 计算质心
library(terra)
library(tidyterra)
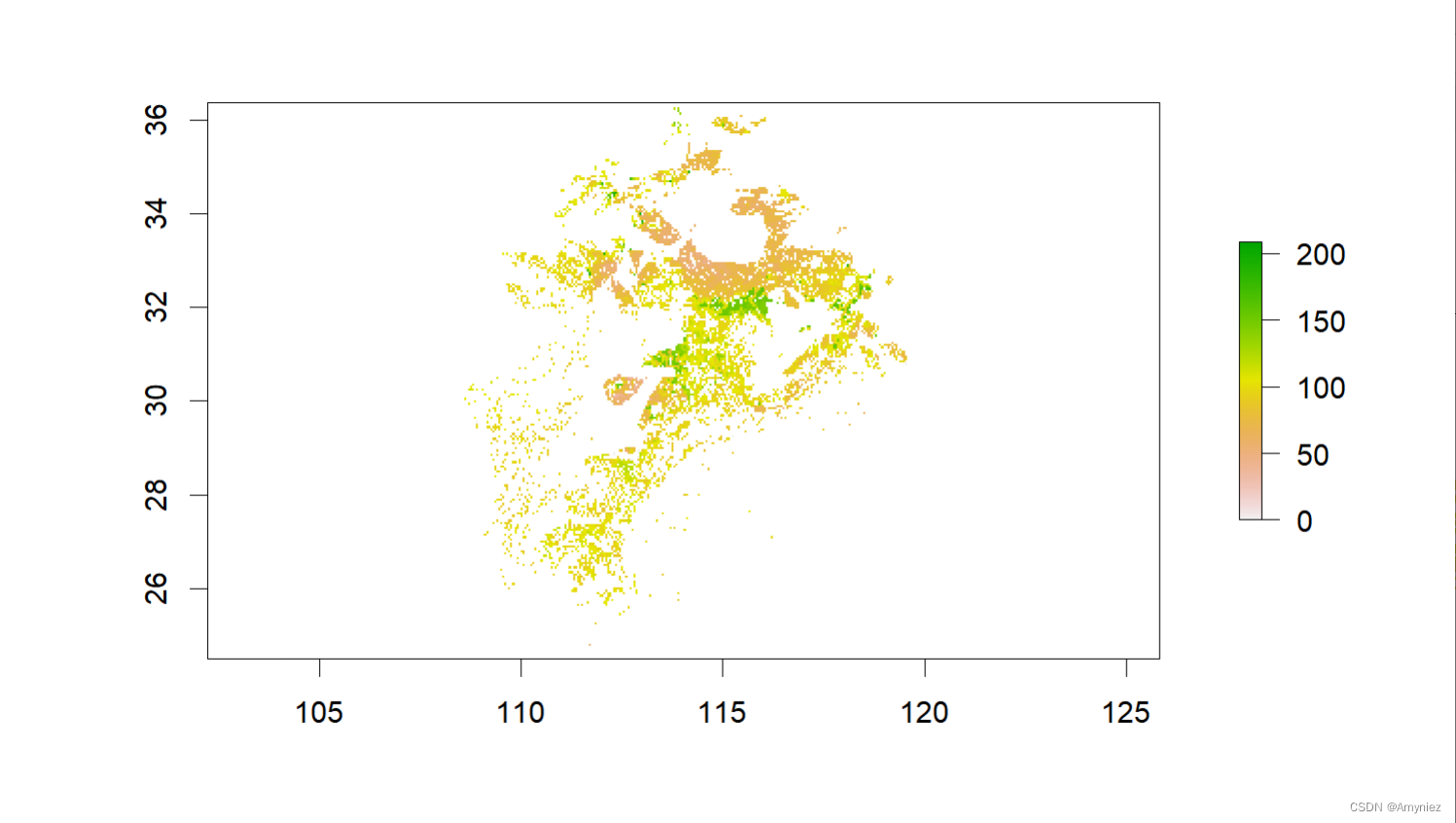
library(ggplot2)bj_dem <- raster("D:/Datasets/w001001.adf")
plot(bj_dem)(sp_sf <- bj_dem %>% calc(x = .,fun = function(x) ifelse(x < 100,x,NA)) %>% # 按属性筛选rasterToPolygons() %>% st_as_sf() %>% summarise(geometry = st_union(geometry)) %>% st_make_valid())
plot(sp_sf)

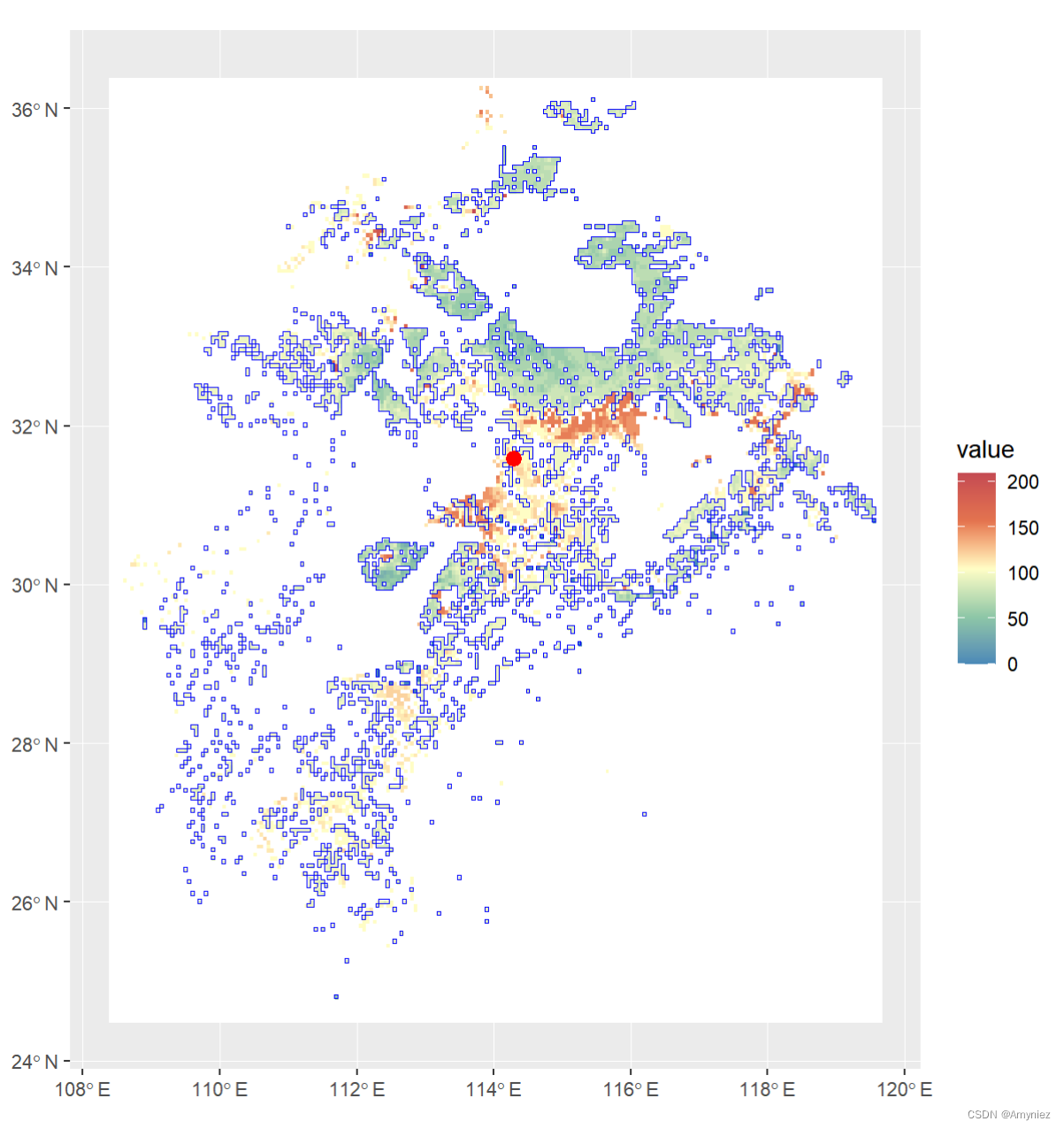
centroid <- st_centroid(sp_sf)ggplot()+geom_spatraster(data = rast(bj_dem)) +scale_fill_whitebox_c(palette = "muted",na.value = "white")+geom_sf(data = sp_sf,alpha = 0,color = "blue")+geom_sf(data = centroid,size = 3,color = "red")
4. 完整案例
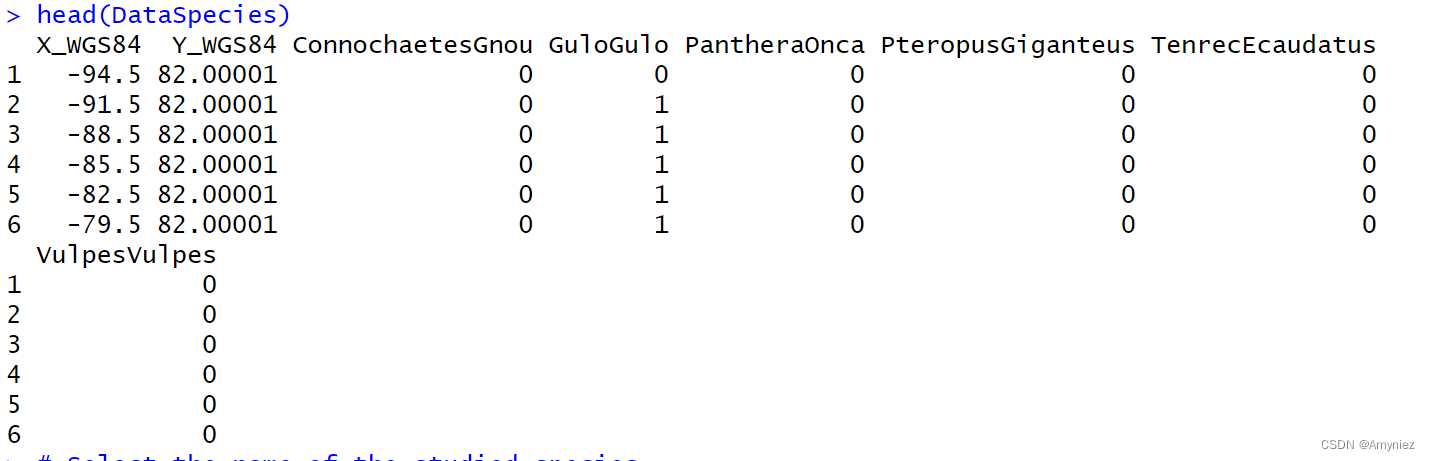
# Load species occurrences (6 species available)
data(DataSpecies)
head(DataSpecies)# Select the name of the studied species
myRespName <- 'GuloGulo'# Get corresponding presence/absence data
myResp <- as.numeric(DataSpecies[, myRespName])# Get corresponding XY coordinates
myRespXY <- DataSpecies[, c('X_WGS84', 'Y_WGS84')]# Load environmental variables extracted from BIOCLIM (bio_3, bio_4, bio_7, bio_11 & bio_12)
data(bioclim_current)
myExpl <- terra::rast(bioclim_current)## --------------------------------------------------------------------------------
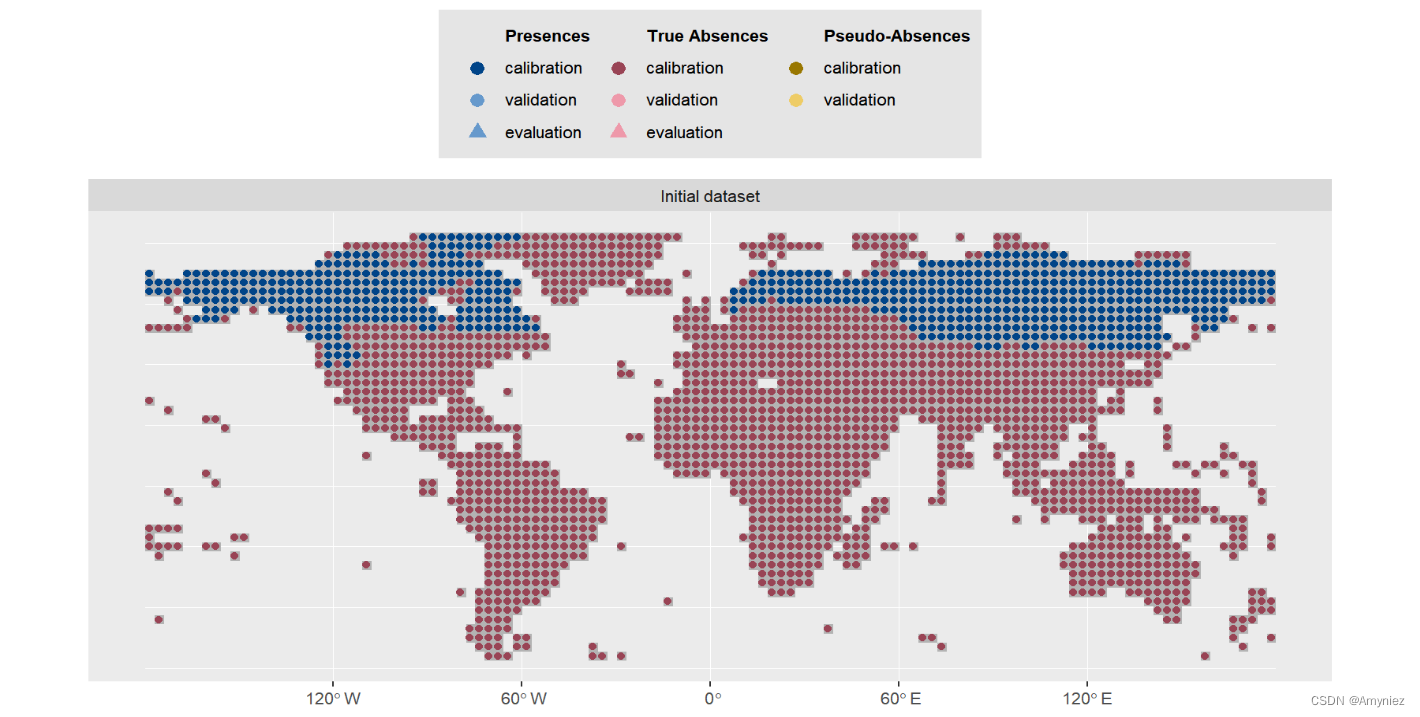
# Format Data with true absences
myBiomodData <- BIOMOD_FormatingData(resp.var = myResp,expl.var = myExpl,resp.xy = myRespXY,resp.name = myRespName)
myBiomodData
summary(myBiomodData)
plot(myBiomodData)
物种分布数据: